PCR和RT-PCR
Ø 聚合酶链式反应
l 定义:PCR是一种能够在短时间内将单个DNA分子扩增数百万倍的生化过程。
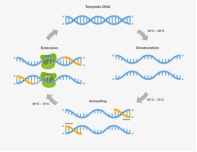
l 过程:包括三个连续步骤
(1) 变性,对双链DNA模板进行加热,使其解离;
(2) 退火,被称为引物的短DNA分子与目标DNA的侧翼区域结合;
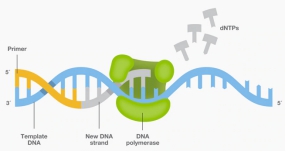
(3) 延伸,DNA聚合酶沿着模板链将引物3’端进行延伸。
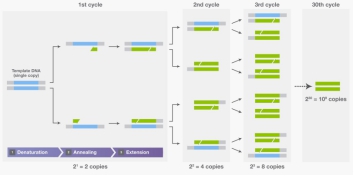
将这些步骤重复(“循环”)25-35次,即可按指数方式获得精确的目标DNA拷贝(如图所示)
l 所需条件及主要参数
条件:Primers、DNA、DNA Polymerase、Buffer、dNTP、dH2O (具体比例参见实验Protocol)
参数:PCR循环数:通常为25-35次。不建议使用超过45个循环,循环数过多将导致非特异性条带产生。
Tm值:Tm=( 2℃ × number of A , T ) + ( 3℃ × number of G , C )
延伸时间:以实验室常用酶Taq酶为例,产物长度每1kb需要1min,可适当延长3-5 s。
※模板量不超过体系的20%
l 实验室现用几种后续可再补充更正)
DNA聚合酶 | 品牌 | 特点 | 用途 |
Taq DNA Polymerase | Vazmy | 价格便宜,效率高 | 全体普通PCR |
PrimerSTAR | Takara | 高保真 | 构克隆 |
Q5 High-Fidelity PCR | NEB | 高保真 | 点突变 |
l 可能导致后续可再补充更正)
1、 现象:没有产物
可能原因:模板cDNA用量偏低 / Tm温度偏低 / 延伸时间短于产物所需时间
/ 丰度太低 / 酶有问题(这个不太可能,实验室现有几种酶都挺好用)
2、 现象:胶图条带弥散
可能原因:模板cDNA用量过高 / 弥散位置位于100bp大小左右则为引物二聚体,引物有问题
两条引物用量不等(没有提前配好Mix)
3、 现象:胶图条带拖尾(类似微笑、U)
可能原因:模板cDNA用量过高 / 酶的用量过高 / 跑胶电压过高
4、 现象:出现非特异性扩增条带
可能原因:引物特异性不高 / 循环次数过高 / Tm温度不合适
5、实验室现有DPN1酶可能有问题,需确认(有两家的NEB和Thermo),点突变完剩下原载体
Ø 定量即时聚合酶链锁反应(
l 定义:Real Time PCR是一种做为常规PCR的衍生反应,以荧光染剂侦测每次PCR循环后产物总量的方法技术。主要是通过荧光信号的变化实时监测PCR扩增反应中每一个循环扩增产物量的变化,通过ct值和标准曲线的关系对起始模板进行定量分析。
l 主要参数
基线(baseline):第 3-15 个循环的荧光值就是基线(baseline),是由测量的偶然误差引起的。
阈值(threshold):指在扩增曲线的指数增长区域内的适当位置上设定的荧光检出界限,一般是基线的标准偏差的 10 倍。
Ct 值:每个反应管里的荧光值达到阈值时的 PCR 循环次数。Ct 值跟初始模板的量成反比。

l 常用方法及原理
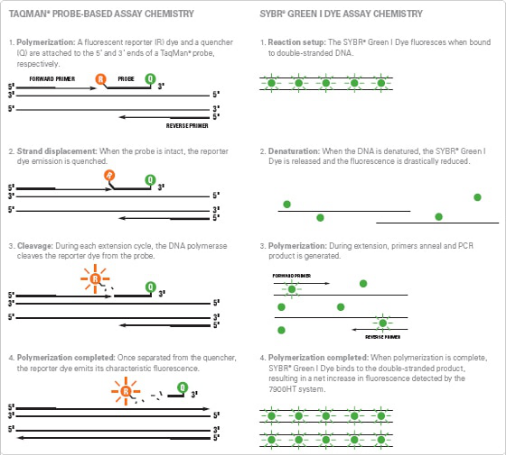
、荧光染料嵌合法
SYBR 染料通过与 PCR 过程中形成的双链 DNA 结合来检测聚合物链反应 (PCR) 产物。
SYBR Green是一种荧光染料,结合于所有dsDNA双螺旋小沟区域的具有绿色激发波长的染料。在游离状态下,SYBR Green发出微弱的荧光,一旦与双链DNA结合后,荧光大大增强。因此,SYBR Green的荧光信号强度与双链DNA的数量相关,可以根据荧光信号检测出PCR体系存在的双链DNA数量。
、荧光探针法(
构建一段寡核苷酸探针:5’端标记报告基团荧光染料,3’ 端标记淬灭基团染料。当探针完整时,邻位的淬灭基团可以通过荧光共振能量转移 (FRET) 大幅降低报告基团染料发出的荧光。
如果存在目标序列,探针便会在其中一个引物结合位点的下游发生退火,并随着引物的延伸通过Taq DNA 聚合酶的 5’核酸酶活性,完成切除。
探针的切除将会引起:
-- 将报告基团染料和淬灭基团染料进行分离,增强了报告基团染料的信号。
-- 将探针从目的链上去除,使引物继续沿模板链末端延伸。因此,探针的介入并不会抑制整个PCR过程。
每经过一个循环,就会有更多的报告基因染料分子从各自的探针上切断,荧光强度会随着合成的扩增片段数量的增加而增加。
基于 SYBR Green 的检测 | 基于 TaqMan 的检测 | |
特异性 | 中 | 高 |
灵敏度低的拷贝数 | 可变的 | 1-10 拷贝数 |
可重复 | 中 | 高 |
多重性 |
| ✔ |
预设计实验 |
| ✔ |
通常需要用户设计、 实验优化 | ✔ |
|
基因表达 | 低等水平定量 | 高等水平定量 |
应用 | ||
DNA 定量(病原体检测) | DNA 定量 | |
| ||
多重 |

l 以
1、按照体系配置预混液,充分涡旋混匀。(一个样品需3个复孔)
| 1个孔 | N个孔 |
TB Green Premix Ex Taq II | 5.0 | 以N+2总量 配置MIX |
Primer-For | 0.4 | |
Primer-Rev | 0.4 | |
ddH2O | 2.2 | |
cDNA | 2.0 | 注:逆转录后cDNA需稀释10倍取用,定量18s需稀释300倍 |
Total | 10μL |
|
2、取96孔 / 384孔板,按设计分别加入Mix和cDNA。
注意:全程冰上操作。TB Green Premix Ex Taq II需避光溶解,并且尽量加快上样速度,缩短曝光时间。
3、加样完毕后,贴上封口膜。用牛皮纸光滑面覆盖贴膜,反复摁压紧实。尽量避免接触封口膜,防止留下污渍。
4、离心。注意放置方向。
5、上机检测。注意预约时间,避免挤占他人时间。
6、拷回数据时,建议将板子取回,抽样跑胶验证条带是否特异。
※那个提取组织RNA试剂盒,里面虽然有去除基因组DNA的操作,但是逆转录前还是得经过TAKARA的去基因组操作,不然后续定量有问题。
l 上机程序(都在平台电脑里,这里只是做个参考)
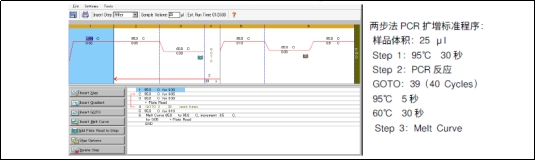
1、 CFX96 Real Time PCR Detection System
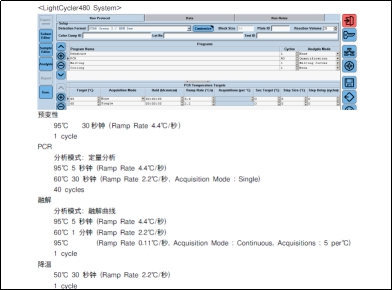
2、LightCycler /LightCycler 480 System (罗氏,可用384孔板)
l 结果分析
1、熔解曲线
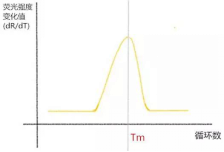
 熔解曲线就是随温度升高DNA的双螺旋结构降解程度的曲线。扩增反应完成后,通过逐渐增加温度同时监测每一步的荧光信号来产生熔解曲线。熔解温度上有一特征峰(Tm,DNA双链解链50%的温度),用这个特征峰就可以将特异产物与其它产物如引物二聚体区分开,因为它们在不同的温度熔解度不一样。用荧光信号改变的负的一次导数(dR/dT)与温度作图,得出熔解曲线。曲线横坐标是温度,纵坐标是荧光强度的变化值。
熔解曲线就是随温度升高DNA的双螺旋结构降解程度的曲线。扩增反应完成后,通过逐渐增加温度同时监测每一步的荧光信号来产生熔解曲线。熔解温度上有一特征峰(Tm,DNA双链解链50%的温度),用这个特征峰就可以将特异产物与其它产物如引物二聚体区分开,因为它们在不同的温度熔解度不一样。用荧光信号改变的负的一次导数(dR/dT)与温度作图,得出熔解曲线。曲线横坐标是温度,纵坐标是荧光强度的变化值。
SYBR Green法进行检测时,可根据熔解曲线确认PCR产物的特异性。正常情况下,荧光定量PCR的产物是特异的,这时的熔解曲线为单峰。(如图)
2、相对定量分析
相对定量用于测定单个样品基因的差异表达分析或者比较两个或两个以上样品中某个基因表达量的变化,则其结果必须用内参基因(管家基因)校正。管家基因通常指维持细胞基本代谢活动所必须的基因,如GAPDH/β-Actin/18s rRNA等,实际操作中可依据文献或具体实验进行筛选。
实验室群里有老师设计的分析模板,可自行下载。
l 实验可能遇到的问题及注意事项(后续可再补充更正)
1、三个复孔间的CT值误差大于0.01?
答:1)配置混样后没有充分混匀,导致三个复孔间会出现差异。
2)引物设计缺乏特异性或是引物与模板不匹配,导致复孔的结果出现差异;
3)加样时,移液枪操作引入误差。
2、为什么内参的CT值过高?
答:1)先确定你的引物是否正确,也就是排除引物的问题。
2)RNA的抽提时出现了降解导致了RNA片段的不完整,重新抽提RNA并反转。
3)实验操作慢/放置太久,导致SYBR曝光时间过长,整体CT值升高。
4)模板放置过久,发生降解。
3、为什么熔解曲线有双峰?
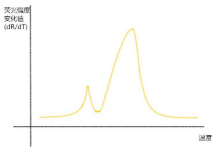
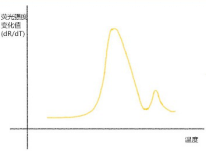
1)正常情况:引物特异性好
2)前置杂峰:1、短于目的产物的杂带;2、引物二聚体;
3)后置杂峰:1、长于目的产物的杂带
4、内参基因使用:惯用18S-rRNA,GAPDH跟分化相关,我们实验室不推荐用。如果课题没有跟细胞骨架明确关系,可以用ACTIN。
5、细节建议: cDNA可由20ul稀释10倍,然后取2ul进行pcr。这个量对大多数基因都好,丰度低的基因,可取到最大量,如6ul。尽量避免取1ul进行pcr,准确度下降。对18s,取2-4ul稀释后的cDNA继续稀释,避免取1ul再稀释。
注意PCR酶和突变用酶的存放。特别是进行突变的酶,用量少、使用时间长但频次多,建议使用时现拿出、取完直接放回-20。若买的包装较大,建议进行必要的分装,如:分装为2份;但不可过度分装。
Ø 引物设计
l 引物设计原则
1. 引物3’端最后一个碱基最好为G或者C;
2. 引物3’端最后8个碱基应避免出现连续错配;
3. 引物3’端应避免出现发夹结构;
4. 正向引物和反向引物的Tm值相差不超过1℃为佳,Tm值调整至55-65℃为佳(计算引物Tm值推荐用软件);
5. 引物额外附加序列,即与模板非配对序列,不应参与引物Tm值计算;
6. 引物的GC含量控制在40-60%之间;
7. 引物A、G、C、T整体分布要尽量均匀,避免使用GC或者AT含量高的区域;
8. 引物单链长度建议在17-24bp为佳;
9. 避开引物内部或者两条引物之间有5个碱基以上的互补序列,两条引物的3’端避免有3个碱基以上的互补序列;
10. 引物设计完毕请使用NCBI BLAST功能检索引物特异性,以避免非特异性扩增产生,
※ 如果是作为RT-PCR引物设计,产物长度建议控制在100-250bp之间。
l 普通
方法一:工具设计
1、 利用在线工具
NCBI的Primer-Blast(http://www.ncbi.nlm.nih.gov/tools/primer-blast/)
Primer3 Plus (http://www.primer3plus.com/)
2、 下载引物设计软件
PrimerPrimer系列
Oligo6(可对引物评价打分)
方法二、手动设计
许多特殊引物(例如circRNA所用)需自行设计,可直接在SnapGene中选取合适位置设计引物,按照基本设计原则,调整好长度和Tm值。设计完毕后去网站中检验特异性。
l 酶切引物设计
1、普通引物设计好后,分别在两条引物的5’端引入所需的酶切位点序列。
2、在引物的5’端额外加上两个保护碱基(网上有内切酶相应的保护碱基推荐,设计时可进行查阅)
Primer-For:5‘-保护碱基+酶切位点+目的基因序列-3‘
Primer-Rev:5‘-保护碱基+酶切位点+目的基因序列-3‘
l 基因定量引物设计(可用工具)
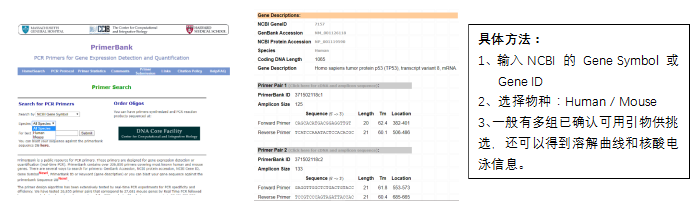
PrimerBank(PCR Primers for Gene Expression Detection and Quantification)
网址:https://pga.mgh.harvard.edu/primerbank/
l 手动设计
设计方法同普通PCR引物大体相似,特别注意的是:1、产物长度建议控制在100-250bp之间,且两条引物的Tm值尽量接近一致;2、尽量在Exon Junction 上设计引物,限制基因组DNA扩增;3、严格的引物特异性检验。不仅在网上Blast比对分析,还需PCR跑胶确定条带为特异单一。
另外,在完成RT-PCR实验后,还需确认引物特异性。
1、配合PCR产物熔解曲线的分析:如果熔解曲线得到单一峰,一般认为无非特异性扩増,用该方法定量准确;如果熔解曲线岀现杂峰,提示以此定量不准确。
2、将RT-PCR产物跑胶观察,条带单一方定量准确,若出现非特异性杂带,则定量不准确。
l 检验引物特异性(可用工具)
、
网址:https://www.ncbi.nlm.nih.gov/tools/primer-blast/index.cgi?LINK_LOC=BlastHome

注意:看引物对非目标基因的检测潜力,如果引物对于非目标基因扩出的产物长度与目标产物长度大小接近,这个引物的非特异性就很强,不建议使用。
、核酸电泳检验
新引物(尤其是定量引物)设计完后,务必进行PCR跑胶检测,是否条带为特异单一,若非特异,则重新设计。
l 实验室常用内参引物序列(后续可再更新补充)
18s rRNA /β-Actin
18S(qPCR)-F | GACTCAACACGGGAAACCTC |
18S(qPCR)-R | AGCATGCCAGAGTCTCGTTC |




